भारतीय वैज्ञानिकों ने 50 वर्ष पुराने जैविक नियम को नए सिरे से लिखने में मदद की
एक नए अध्ययन ने जीवाणु जीन विनियमन के एक केन्द्रीय पाठ्यपुस्तक मॉडल को उलट दिया है और जीवाणु जीन विनियमन एवं उसके विकास को समझने के नए रास्ते खोले हैं। इससे संक्रमण तंत्र को अवरुद्ध करने वाले बेहतर एंटीबायोटिक्स या नियामक अवरोधकों को डिजाइन करने में मदद मिल सकती है।
बैक्टीरिया अपने जीन को कैसे नियंत्रित करते हैं, इसकी समझ सूक्ष्मजीव द्वारा तनाव पर प्रतिक्रिया देने के तरीकों से लेकर हमारे द्वारा उन्हें रोकने हेतु एंटीबायोटिक्स को डिजाइन करने के तरीकों तक से संबंधित हमारी तमाम अवधारणाओं को प्रभावित करती है। यदि विभिन्न प्रजातियों में जीन विनियमन की मूल क्रियाविधि भिन्न होती है, तो इसका अर्थ संक्रमणों से लड़ने के लिए नई रणनीतियां या उपयोगी यौगिक बनाने के लिए बैक्टीरिया का उपयोग भी हो सकता है।
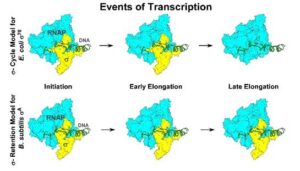
लगभग 50 वर्षों से, जीव विज्ञान इस कहानी को बताता आ रहा है कि कैसे बैक्टीरिया तथाकथित “σ (सिग्मा) चक्र” की सहायता से अपने जीन को सक्रिय करते हैं – ये वे कारक हैं जो आरएनए बहुलकों (पॉलीमरेज) को बांधकर प्रतिलेखन शुरू करते हैं और फिर विस्तार को संभव बनाने के लिए अलग हो जाते हैं। यह अवधारणा मुख्यतः बैक्टीरिया के स्ट्रेन ई. कोलाई σ70 के अवलोकनों पर आधारित है।
हालांकि, विज्ञान एवं प्रौद्योगिकी विभाग (डीएसटी) के स्वायत्त संस्थान बोस इंस्टीट्यूट और रटगर्स विश्वविद्यालय के शोधकर्ताओं ने बताया है कि यह चक्र एक सार्वभौमिक घटना नहीं है।
प्रोसीडिंग्स ऑफ द नेशनल एकेडमी ऑफ साइंसेज (पीएनएएस) में प्रकाशित एक अध्ययन में उन्होंने बताया है कि, दशकों के वैज्ञानिक विश्वास के उलट, बैसिलस सबटिलिस – σए – में प्रमुख प्रतिलेखन आरंभ कारक और एस्चेरिचिया कोली σ70 कारक का एक संशोधित संस्करण, प्रतिलेखन के आरंभ होने के बाद अलग होने के बजाय, इस पूरी प्रक्रिया के दौरान आरएनए बहुलकों (पॉलीमरेज) से बंधा रहता है।
बोस संस्थान के लेखक डॉ. जयंत मुखोपाध्याय ने बताया, “हमारा शोध यह दर्शाता है कि बैसिलस सबटिलिस में, σA कारक प्रतिलेखन की प्रक्रिया के दौरान आरएनए बहुलकों (पॉलीमरेज) से जुड़ा रहता है।” उन्होंने आगे कहा, “इससे बैक्टीरिया प्रतिलेखन और जीन विनियमन के बारे में हमारी सोच में बुनियादी बदलाव आता है।”
जैव रासायनिक परख, क्रोमेटिन इम्यूनोप्रीसिपिटेशन और प्रतिदीप्ति-आधारित इमेजिंग जैसी आधुनिक तकनीकों के संयोजन का उपयोग करते हुए, शोधकर्ताओं ने सिग्मा कारक के व्यवहार को वास्तविक समय में देखा। उन्होंने पाया कि बैसिलस सबटिलिस σ ए और ई. कोलाई σ70 का एक प्रकार, जिसमें 1.1 नामक भाग नहीं होता है, ट्रांसक्रिप्शन कॉम्प्लेक्स के साथ स्थिर रूप से जुड़े रहते हैं। यह पूर्ण लंबाई वाले ई. कोलाई σ70 के बिल्कुल विपरीत है, जो विस्तार के दौरान यादृच्छिक रूप से अलग हो जाता है।
बोस इंस्टीट्यूट के सह-लेखक अनिरुद्ध तिवारी ने कहा, “ये निष्कर्ष इस बात का ठोस प्रमाण देते हैं कि लंबे समय से स्वीकृत σ चक्र सभी बैक्टीरिया पर लागू नहीं होता। यह बैक्टीरिया के जीन नियमन और उसके विकास को समझने के नए रास्ते खोलता है।”
इस खोज के सूक्ष्म जीव विज्ञान पर व्यापक प्रभाव होंगे तथा यह संभावित रूप से शोधकर्ताओं के जीवाणु शरीरक्रिया विज्ञान, तनाव प्रतिक्रिया तथा प्रतिलेखन को लक्षित करने वाले एंटीबायोटिक्स के विकास के लिए कार्य करने के तौर-तरीकों को प्रभावित करेगा।
जीन विनियमन को नियंत्रित करके, वैज्ञानिक ऐसे सूक्ष्मजीवों को डिजाइन कर सकते हैं जो जैव ईंधन, जैविक रूप से अपघटित होने वाले प्लास्टिक या चिकित्सीय यौगिकों का कुशलतापूर्वक उत्पादन कर सकते हैं।
डॉ. तिवारी और डॉ. मुखोपाध्याय के अलावा, कोलकाता स्थित बोस इंस्टीट्यूट से श्रेया सेनगुप्ता, सौम्या मुखर्जी, नीलांजना हाजरा और संयुक्त राज्य अमेरिका के रटगर्स यूनिवर्सिटी से योन डब्ल्यू. एब्राइट, रिचर्ड एच. एब्राइट ने इस अध्ययन में योगदान दिया।
——————————————————————————————–
प्रकाशन लिंक: doi:10.1073/pnas.2503801122

